Code
library(tidyverse)
library(ggpubr)
library(qqplotr)Diseño experimental
library(tidyverse)
library(ggpubr)
library(qqplotr)
datos <- read_csv("datos/lecheria_eeuu.csv")
datos |> head()
ggqqplot(datos$avg_price_milk)
datos |>
select(-year) |>
pivot_longer(cols = everything()) |>
ggplot(aes(sample = value)) +
facet_wrap(~name, scales = "free") +
stat_qq_band() +
stat_qq_line() +
stat_qq_point()
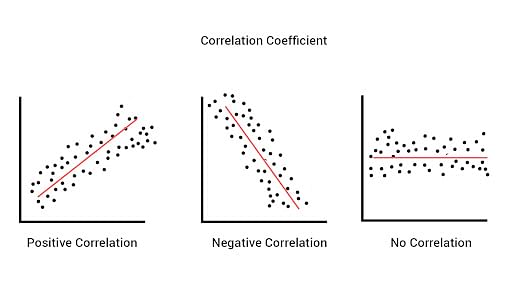
\[\rho_{(X,Y)} = \frac{Cov_{(X,Y)}}{\sigma_X\times\sigma_Y} = \frac{\sum_{i=1}^{n}(X_i-\mu_X)(Y_i-\mu_Y)}{\sigma_X\times\sigma_Y}\]
covarianza <- cov(x = datos$dairy_ration, y = datos$avg_price_milk)
desv_x <- sd(datos$dairy_ration)
desv_y <- sd(datos$avg_price_milk)
covarianza / (desv_x * desv_y)[1] 0.8172358cor(x = datos$dairy_ration,
y = datos$avg_price_milk)[1] 0.8172358\[\rho = 1 - \frac{6\sum D^2}{N (N^2 - 1)}\]
cor(x = datos$dairy_ration,
y = datos$avg_price_milk,
method = "spearman")[1] 0.63335datos |>
ggplot(aes(x = dairy_ration, y = avg_price_milk)) +
geom_point()
datos |>
ggplot(aes(x = dairy_ration, y = avg_price_milk)) +
geom_point() +
geom_smooth(method = "lm")
datos |>
ggplot(aes(x = dairy_ration, y = avg_price_milk)) +
geom_point() +
geom_smooth()
\[Y_i = \beta_0\ +\ \beta_1X\] \[\hat{Y_i} = \hat{\beta_0}\ + \hat{\beta_1}X_i\ + \hat{\epsilon_i}\]
\[Y_i = E(Y|X_i)\ +\ \epsilon_i\\\]
\[Y_i = E(Y|X_i)\ +\ \epsilon_i\\ Y_i = \beta_0 +\ \beta_1X_i\ +\ \epsilon_i\]
modelo1 <- lm(avg_price_milk ~ dairy_ration, data = datos)
modelo1 |> summary()
Call:
lm(formula = avg_price_milk ~ dairy_ration, data = datos)
Residuals:
Min 1Q Median 3Q Max
-0.033635 -0.008394 -0.002785 0.003106 0.055094
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 0.086176 0.007885 10.930 1.67e-12 ***
dairy_ration 1.038172 0.127443 8.146 2.10e-09 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 0.01655 on 33 degrees of freedom
Multiple R-squared: 0.6679, Adjusted R-squared: 0.6578
F-statistic: 66.36 on 1 and 33 DF, p-value: 2.102e-09\[\hat{y} = 0.086176 + (1.038172 \times X)\]
0.086176 + (1.038172 * 0.082)[1] 0.1713061datos_test <-
datos |>
sample_n(size = 10) |>
select(dairy_ration)
datos_testpredict(object = modelo1,
newdata = datos_test) 1 2 3 4 5 6 7 8
0.2077383 0.1315572 0.1384699 0.1373187 0.1978416 0.2123142 0.1384508 0.1360335
9 10
0.1849064 0.1445377 \[R^2 = \rho^2\]
correlacion_pearson <- 0.8172358
r2 <- correlacion_pearson ^ 2
r2[1] 0.6678744\[R^2_a = 1 - \frac{N-1}{N-k-1} (1 - R^2)\]
N <- nrow(datos) # número de datos
k <- 1 # pendiente (parámetro Beta1)
1 - ((N - 1) / (N - k - 1)) * (1 - r2)[1] 0.6578099\[RMSE = \sqrt{\frac{\sum_{i = 1}^{n} (y - \hat{y})^2}{N}}\]
valores_reales <- datos$avg_price_milk # variable y
predichos_m1 <- modelo1$fitted.values
diferencia_cuadrado <- (valores_reales - predichos_m1)^2
suma_cuadrados <- sum(diferencia_cuadrado)
metrica_rmse <- sqrt(suma_cuadrados / N)
metrica_rmse[1] 0.01606818library(yardstick)
rmse_vec(truth = valores_reales, estimate = predichos_m1)[1] 0.01606818\[AIC = -2ln(L) + k\times n_{par}\]
log_l <- logLik(modelo1) # log verosimilitud
n_par <- length(coefficients(modelo1)) # número de parámetros
-2 * log_l + 2 * n_par'log Lik.' -185.8383 (df=3)AIC(modelo1)[1] -183.8383
residuales <- modelo1$residuals
residuales 1 2 3 4 5
-0.0063963730 -0.0004507765 0.0039146871 -0.0045249485 -0.0066020776
6 7 8 9 10
-0.0058987810 -0.0003646907 0.0030543723 -0.0131944037 -0.0027847996
11 12 13 14 15
0.0004026023 -0.0107404425 -0.0005572382 -0.0059314158 -0.0073187107
16 17 18 19 20
-0.0101901072 -0.0010347143 -0.0115376727 0.0197303289 0.0161825420
21 22 23 24 25
-0.0051450271 0.0180051551 -0.0137001647 -0.0110334541 0.0208318798
26 27 28 29 30
0.0162116339 -0.0094699308 0.0342720367 0.0016622358 -0.0336354964
31 32 33 34 35
0.0020442379 0.0031583847 -0.0273141708 -0.0067382644 0.0550935636 valores_reales - predichos_m1 1 2 3 4 5
-0.0063963730 -0.0004507765 0.0039146871 -0.0045249485 -0.0066020776
6 7 8 9 10
-0.0058987810 -0.0003646907 0.0030543723 -0.0131944037 -0.0027847996
11 12 13 14 15
0.0004026023 -0.0107404425 -0.0005572382 -0.0059314158 -0.0073187107
16 17 18 19 20
-0.0101901072 -0.0010347143 -0.0115376727 0.0197303289 0.0161825420
21 22 23 24 25
-0.0051450271 0.0180051551 -0.0137001647 -0.0110334541 0.0208318798
26 27 28 29 30
0.0162116339 -0.0094699308 0.0342720367 0.0016622358 -0.0336354964
31 32 33 34 35
0.0020442379 0.0031583847 -0.0273141708 -0.0067382644 0.0550935636 # residuales |> hist()
residuales |>
enframe() |>
ggplot(aes(x = value)) +
geom_histogram(bins = 7, color = "black")
ggqqplot(residuales)
par(mfrow = c(2, 2)) # cuatro gráficos
modelo1 |>
plot()
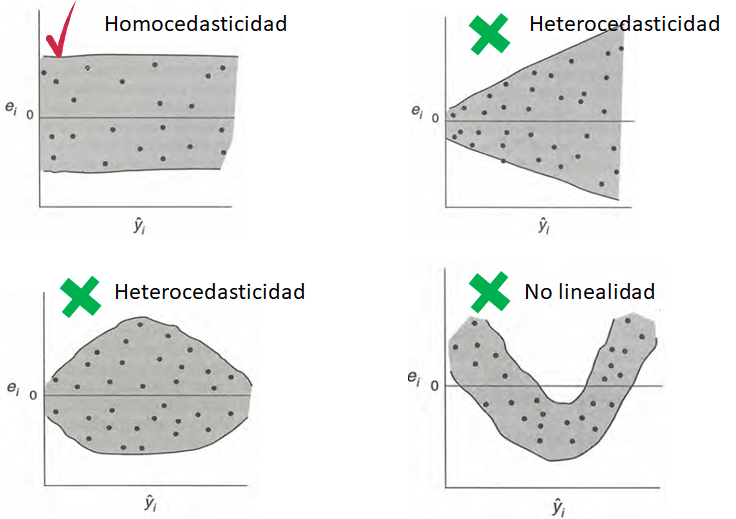
data_residuales <-
data.frame(predichos = predichos_m1, residuales = residuales)
data_residuales |>
ggplot(aes(x = predichos, y = residuales)) +
geom_point() +
geom_hline(yintercept = 0, color = "red", linetype = 2) +
geom_smooth(se = FALSE)
datos |>
pivot_longer(cols = -avg_price_milk) |>
ggplot(aes(x = value, y = avg_price_milk)) +
facet_wrap(~name, ncol = 3, scales = "free_x") +
geom_point() +
geom_smooth(method = "lm")
modelo2 <- lm(avg_price_milk ~ alfalfa_hay_price, data = datos)
modelo3 <- lm(avg_price_milk ~ milk_feed_price_ratio, data = datos)
modelo4 <- lm(avg_price_milk ~ milk_per_cow, data = datos)AIC(modelo1, modelo2, modelo3, modelo4)predichos_m2 <- modelo2$fitted.values
predichos_m3 <- modelo3$fitted.values
predichos_m4 <- modelo4$fitted.values
rmse_vec(truth = valores_reales, estimate = predichos_m1)[1] 0.01606818rmse_vec(truth = valores_reales, estimate = predichos_m2)[1] 0.01409574rmse_vec(truth = valores_reales, estimate = predichos_m3)[1] 0.02553965rmse_vec(truth = valores_reales, estimate = predichos_m4)[1] 0.0207514ggqqplot(modelo1$residuals)
ggqqplot(modelo2$residuals)
ggqqplot(modelo3$residuals)
ggqqplot(modelo4$residuals)
data.frame(residuales = modelo1$residuals,
predichos = modelo1$fitted.values) |>
ggplot(aes(x = predichos, y = residuales)) +
geom_point() +
geom_hline(yintercept = 0,
color = "red",
linetype = 2) +
geom_smooth(se = FALSE)
data.frame(residuales = modelo2$residuals,
predichos = modelo2$fitted.values) |>
ggplot(aes(x = predichos, y = residuales)) +
geom_point() +
geom_hline(yintercept = 0,
color = "red",
linetype = 2) +
geom_smooth(se = FALSE)
data.frame(residuales = modelo3$residuals,
predichos = modelo3$fitted.values) |>
ggplot(aes(x = predichos, y = residuales)) +
geom_point() +
geom_hline(yintercept = 0,
color = "red",
linetype = 2) +
geom_smooth(se = FALSE)
data.frame(residuales = modelo4$residuals,
predichos = modelo4$fitted.values) |>
ggplot(aes(x = predichos, y = residuales)) +
geom_point() +
geom_hline(yintercept = 0,
color = "red",
linetype = 2) +
geom_smooth(se = FALSE)