Código
library(tidyverse)
library(readxl)
library(infer)
theme_set(theme_bw())Diseño Experimental
library(tidyverse)
library(readxl)
library(infer)
theme_set(theme_bw())datos <- read_excel("datos/datos-encuestas-historia.xlsx")
datos



ggpubr::ggqqplot(datos$promedio_acad)
\[H_0: \mu = 3.5\]
\[H_1: \mu \neq 3.5\]
\[T = \frac{\bar{X} - \mu}{S/\sqrt{n}}\]
x_barra <- mean(datos$promedio_acad, na.rm = TRUE)
mu_referencia <- 3.5
desviacion_muestral <- sd(datos$promedio_acad, na.rm = TRUE)
raiz_n <- sqrt(nrow(datos))\[T = \frac{3.794516 - 3.5}{0.247371/9.643651} = 11.48158\]
(x_barra - mu_referencia) / (desviacion_muestral / raiz_n)[1] 11.48158
Podemos obtener los límites critícos con R:
qt(p = 0.025, df = 92, lower.tail = TRUE)[1] -1.986086qt(p = 0.025, df = 92, lower.tail = FALSE)[1] 1.986086
\[\bar{X} - t_{\alpha/2, n-1} \times \frac{s}{\sqrt{n}}\]
x_barra - (1.986086 * (desviacion_muestral / raiz_n))[1] 3.743571\[\bar{X} + t_{\alpha/2, n-1} \times \frac{s}{\sqrt{n}}\]
x_barra + (1.986086 * (desviacion_muestral / raiz_n))[1] 3.845462pt(q = -11.48158, df = 92, lower.tail = TRUE)[1] 9.287894e-20pt(q = 11.48158, df = 92, lower.tail = FALSE)[1] 9.287894e-209.287894e-20 + 9.287894e-20[1] 1.857579e-19x: la variable sobre la cual estamos haciendo inferencia. En este caso el promedio_académicoalternative: tipo de hipótesis alternativa. En este es una prueba bilateral usamos “two.sided”conf.level: nivel de confianza (1 - nivel de significancia = 1 - 0.05 = 0.95)mu: valor promedio de referencia. En este caso es 3.5t.test(x = datos$promedio_acad,
alternative = "two.sided",
conf.level = 0.95,
mu = 3.5)
One Sample t-test
data: datos$promedio_acad
t = 11.482, df = 92, p-value < 2.2e-16
alternative hypothesis: true mean is not equal to 3.5
95 percent confidence interval:
3.743571 3.845462
sample estimates:
mean of x
3.794516 prueba_t1 <- t.test(
x = datos$promedio_acad,
alternative = "two.sided",
conf.level = 0.95,
mu = 3.5
)
library(broom)
prueba_t1 |> tidy()wilcox.test(
x = datos$promedio_acad,
alternative = "two.sided",
conf.int = TRUE,
conf.level = 0.95,
mu = 3.5
)
Wilcoxon signed rank test with continuity correction
data: datos$promedio_acad
V = 3465.5, p-value = 6.357e-14
alternative hypothesis: true location is not equal to 3.5
95 percent confidence interval:
3.794980 3.884976
sample estimates:
(pseudo)median
3.84006 ejemplo <- c(45, 32.5, 85.3, 74.3, 110, 26.8, 28.6)
set.seed(2024)
remuestreo1 <- sample(ejemplo, size = 100, replace = TRUE)
remuestreo2 <- sample(ejemplo, size = 100, replace = TRUE)
remuestreo3 <- sample(ejemplo, size = 100, replace = TRUE)
remuestreo4 <- sample(ejemplo, size = 100, replace = TRUE)
remuestreo5 <- sample(ejemplo, size = 100, replace = TRUE)
remuestreo6 <- sample(ejemplo, size = 100, replace = TRUE)
remuestreo7 <- sample(ejemplo, size = 100, replace = TRUE)
remuestreo8 <- sample(ejemplo, size = 100, replace = TRUE)
remuestreo9 <- sample(ejemplo, size = 100, replace = TRUE)
remuestreo10 <- sample(ejemplo, size = 100, replace = TRUE)mean(ejemplo)[1] 57.5prom_r1 <- mean(remuestreo1)
prom_r2 <- mean(remuestreo2)
prom_r3 <- mean(remuestreo3)
prom_r4 <- mean(remuestreo4)
prom_r5 <- mean(remuestreo5)
prom_r6 <- mean(remuestreo6)
prom_r7 <- mean(remuestreo7)
prom_r8 <- mean(remuestreo8)
prom_r9 <- mean(remuestreo9)
prom_r10 <- mean(remuestreo10)
vector_promedios <- c(prom_r1, prom_r2, prom_r3, prom_r4, prom_r5,
prom_r6, prom_r7, prom_r8, prom_r9, prom_r10)
promedio_remuestreos <- mean(vector_promedios)
ggplot(mapping = aes(x = vector_promedios)) +
geom_density() +
geom_vline(xintercept = promedio_remuestreos, color = "red")
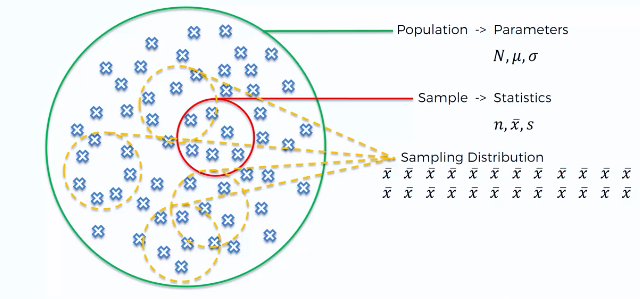
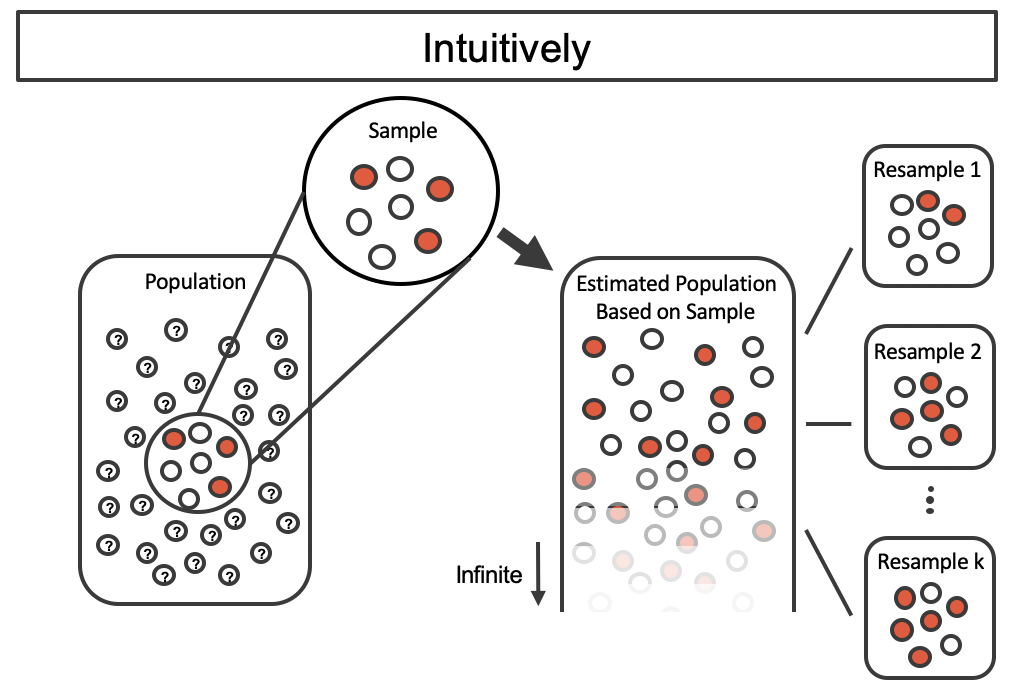
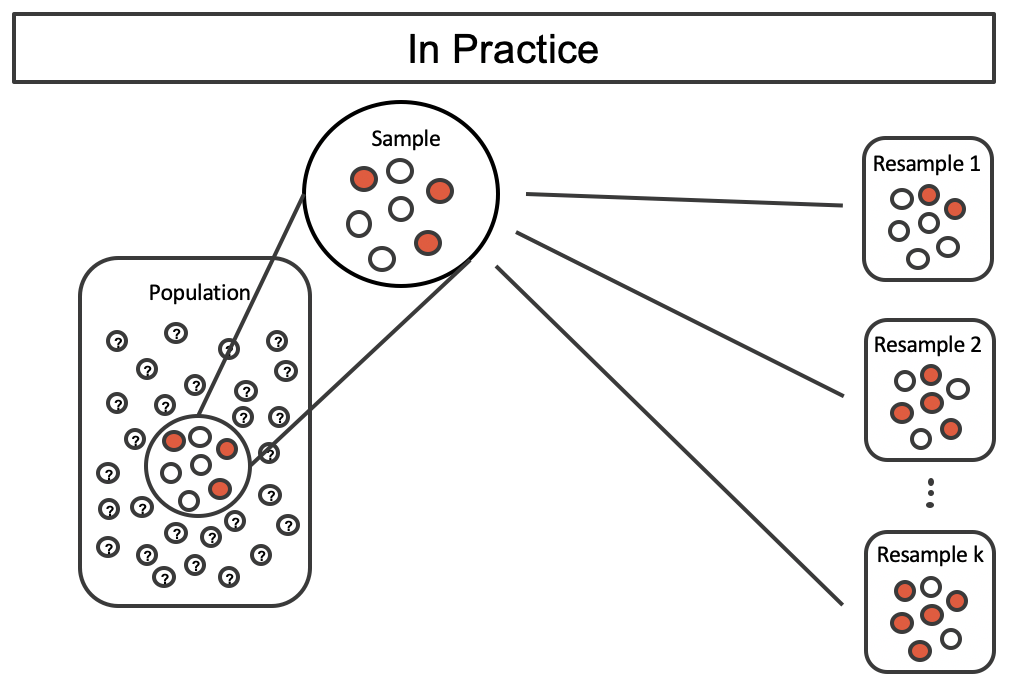
ejemplo <- c(45.67, 67.87, 65, 70, 110, 35.7)
sample(ejemplo, size = 7, replace = TRUE)[1] 45.67 110.00 70.00 45.67 35.70 70.00 45.67inferspecify()generate()calculate()visualize()get_confidence_interval(). Nota: para mejorar la visualización de los intervalos de confianza, se puede utilizar la función shade_confidence_interval()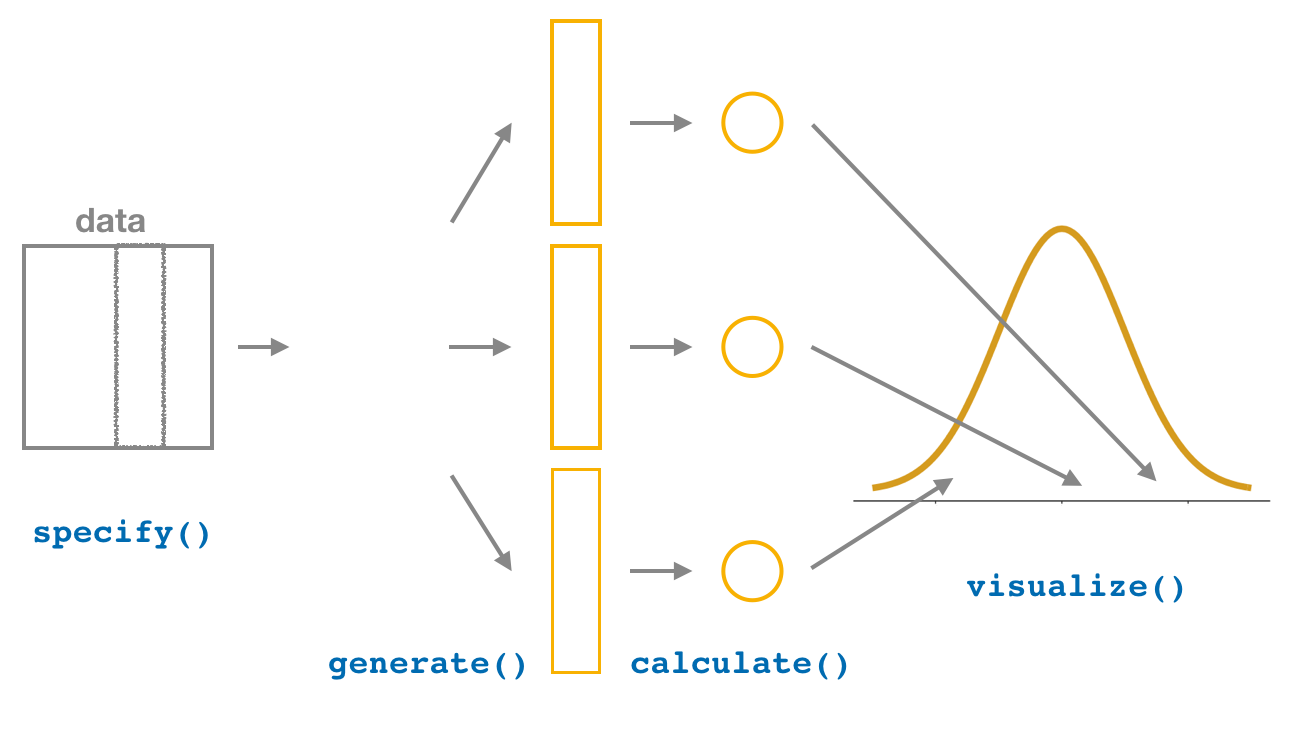
set.seed(2024)
bootstrap_promedio_udea <-
datos |>
specify(response = promedio_acad) |>
generate(reps = 1000, type = "bootstrap") |>
calculate(stat = "mean")
bootstrap_promedio_udeabootstrap_promedio_udea |>
visualize()
 {fig-align=“center” width = “70%”}
{fig-align=“center” width = “70%”}
ic_promedio_percentil <-
bootstrap_promedio_udea |>
get_confidence_interval(level = 0.95, type = "percentile")
ic_promedio_percentilbootstrap_promedio_udea |>
visualize() +
shade_confidence_interval(endpoints = ic_promedio_percentil) +
geom_vline(
xintercept = x_barra,
color = "red",
lty = 2,
size = 1.5
) +
geom_vline(
xintercept = mean(datos$promedio_acad),
color = "black",
lty = 2,
size = 1.5
)
ic_promedio_error_est <-
bootstrap_promedio_udea |>
get_confidence_interval(type = "se", point_estimate = x_barra)
ic_promedio_error_estbootstrap_promedio_udea |>
visualize() +
shade_confidence_interval(endpoints = ic_promedio_error_est) +
geom_vline(
xintercept = x_barra,
color = "red",
lty = 2,
size = 1.5
) +
geom_vline(
xintercept = mean(datos$promedio_acad),
color = "black",
lty = 2,
size = 1.5
)
:::
